実験データの処理¶
研究では日常的に測定したデータをプロットして図を作成します。簡単なデー タと図であれば、Excel等で手動で作成しても良いですが、少しデータを加工 してプロットしたい場合、チマチマ手動で処理するのは面倒です。測定データ をpythonプログラム1つでプロットと作図までできれば、日常の作業が短縮さ れます。いくつかデータをプロットする簡単な例を示します。
データを最大強度で規格化してスタックプロット¶
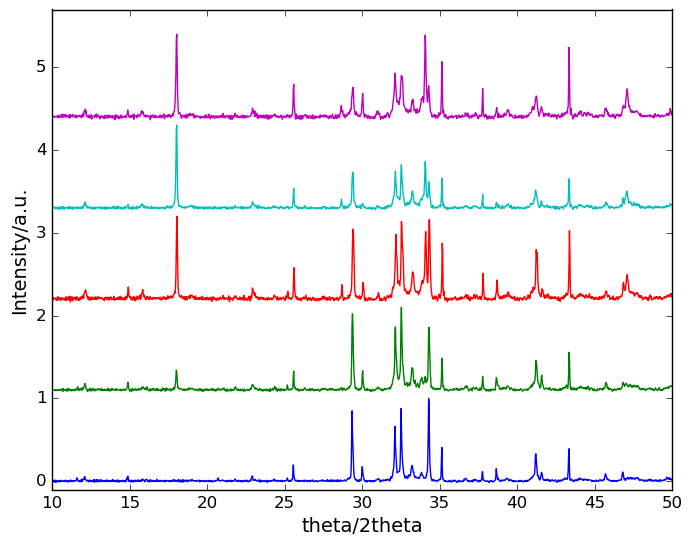
いくつかの試料や条件を変えて測定した分光スペクトルやXRDパターンをスタッ クしてプロットしてみます。データを最大強度1で規格化して、y方向にスタッ クしてプロットします。CUI上から引数で複数のファイルを渡してスタックプ ロットさせます。また、XYのプロット範囲、出力するファイルをargparseを使っ てコマンドオプションで渡すようプログラムしています。
#!/usr/bin/env python
import numpy as np
import matplotlib.pyplot as plt
import argparse
par = argparse.ArgumentParser(description="test")
par.add_argument('files', nargs='+')
par.add_argument('-s', '--yshift', default=1.1, type=float)
par.add_argument('-xl', '--xlim', nargs=2, default=None, type=float)
par.add_argument('-yl', '--ylim', nargs=2, default=None, type=float)
par.add_argument('-o', '--outfig', default=None)
args = par.parse_args()
# 図の体裁 https://matplotlib.org/users/customizing.html
plt.style.use('classic')
plt_dic = {}
plt_dic['legend.fancybox'] = True
plt_dic['legend.labelspacing'] = 0.3
plt_dic['legend.numpoints'] = 3
plt_dic['figure.figsize'] = [8, 6]
plt_dic['font.size'] = 12
plt_dic['legend.fontsize'] = 12
plt_dic['axes.labelsize'] = 14
plt_dic['savefig.bbox'] = 'tight'
plt.rcParams.update(plt_dic)
class DATA():
def __init__(self, infile):
self.infile = infile
self.label = infile
data = np.loadtxt(infile)
self.x = data[:, 0]
self.y = data[:, 1]/np.max(data[:, 1])
data = {}
for f in args.files:
data[f] = DATA(f)
fig, ax = plt.subplots()
for i, d in enumerate(data.values()):
ax.plot(d.x, d.y + i * args.yshift)
ax.set_xlabel('theta/2theta')
ax.set_ylabel('Intensity/a.u.')
if args.ylim is not None:
ax.set_ylim(args.ylim)
if args.xlim is not None:
ax.set_xlim(args.xlim)
if args.outfig is not None:
fig.savefig(args.outfig)
print(args.outfig, "was created.")
# y-ticksの削除
ax.yaxis.set_ticks([])
plt.show()
プログラムの実行は引数でデータの入った複数のファイルを指定します。
stack_plot.py 0h.dat 6h.dat 12h.dat 24h.dat 48h.dat
プロット範囲や出力ファイルを指定すれば、図のファイルを生成できます。
stack_plot.py 0h.dat 6h.dat 12h.dat 24h.dat 48h.dat -o hoge.png -xl 10 50 -yl -0.1 5.7
これで次のようなxrd_stack.pngが生成されます。
測定データのeye guideの線を引く¶
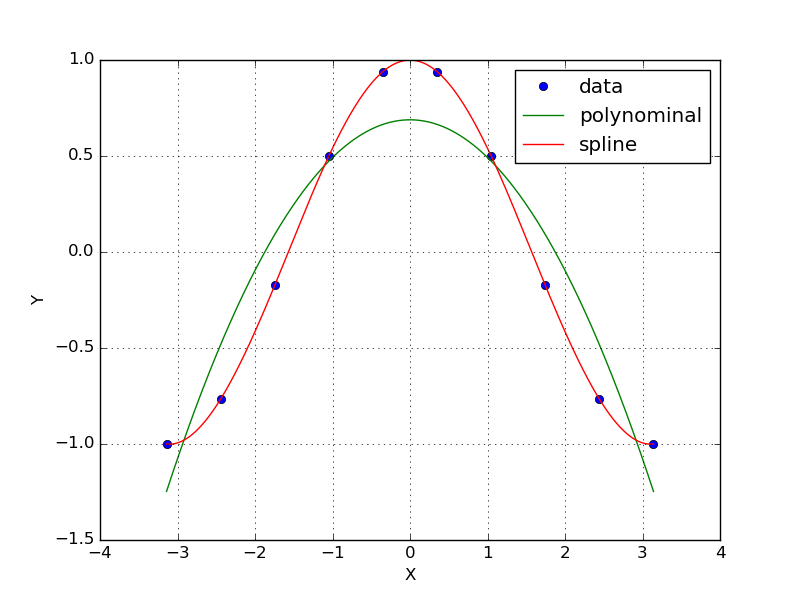
測定データの傾向を示すために、データに近似線を引くことがあります。理論 式があれば、非線形フィッテング等で近似線を引きますが、特に理論式ない場 合は、多項式やスプラインで近似曲線を作ってデータに線を引きます。cosカー ブの適当な測定データを作って近似曲線を作成してみます。
1#!/usr/bin/env python
2import numpy as np
3import matplotlib.pyplot as plt
4from scipy.interpolate import interp1d
5
6x = np.linspace(-np.pi, np.pi, 10)
7y = np.cos(x)
8x_fit = np.linspace(-np.pi, np.pi, 1000)
9
10# https://docs.scipy.org/doc/numpy/reference/generated/numpy.polyfit.html
11# 近似関数は2次関数
12o = np.polyfit(x, y, 3)
13y_poly = np.polyval(o, x_fit)
14
15# https://docs.scipy.org/doc/scipy/reference/generated/scipy.interpolate.interp1d.html
16# cubic spline
17# splineでは必ずデータ点を通る
18o = interp1d(x, y, 'cubic')
19y_spline = o(x_fit)
20
21fig, ax = plt.subplots()
22ax.plot(x, y, "o", label="data")
23ax.plot(x_fit, y_poly, "-", label="polynominal")
24ax.plot(x_fit, y_spline, "-", label="spline")
25ax.legend(loc="best")
26ax.set_xlabel('X')
27ax.set_ylabel('Y')
28fig.savefig("approximate.png")
29plt.show()
実行すると次のようなグラフが得られます。splineは必ずデータ点を通るよう に近似線を作成します。
パラメータ範囲を指定してフィッテング¶
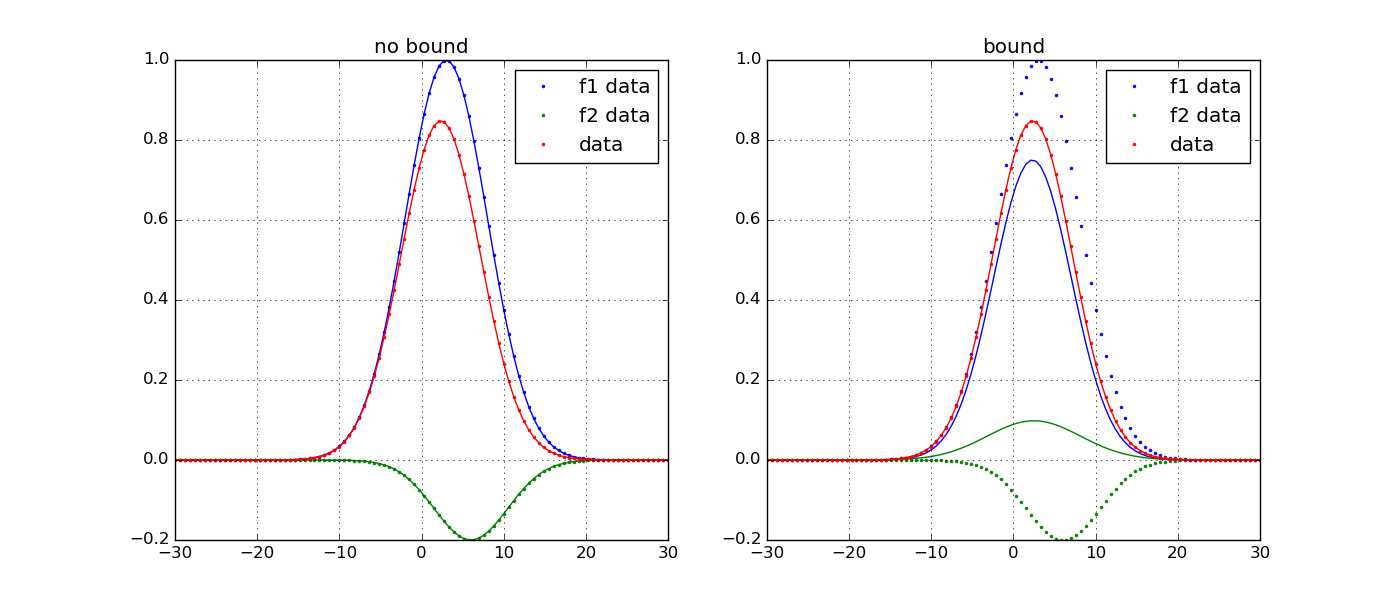
実験的に観測したスペクトルをガウス関数等の釣鐘型の関数でフィッテング (ピーク分離)して面積(成分比)を求めることは、よく行われます。フィッテン グの時に、ガウス関数のパラメータを完全に自由にしてフィッテングすると物 理的におかしいパラメータになることがあります。最適化するパラメータの範 囲を指定することで物理的におかしいパラメータにフィッテングされることを 避けることができます。
パラメータの範囲を指定した場合としない場合でフィッテングするプログラム を以下に示します。モデルデータ(y_exp)は、強度の異なる2つの正負のガウス 関数を足し合わせ作成しています。y_expの範囲制限有り(bound)と無し(no bound)でフィッテングしています。直線がフィッテングで求めたラインで、点 はフィッテングデータです。
#!/usr/bin/env python
import numpy as np
import matplotlib.pyplot as plt
from scipy.optimize import curve_fit
param = np.array([[(0, 1, np.inf), (-10, 3, 10), (0, 50, 100)],
[(0, 1, np.inf), (-10, 6, 10), (0, 10, 100)]])
# 初期値
p0 = param[:, :, 1].reshape(-1)
# 境界値 [[p0_min, p1_min...][p0_max, p1_max...]]
bounds = param[:, :, [0, 2]].reshape((-1, 2)).T
def gauss(x, p):
return p[0] * np.exp(-(x - p[1])**2/p[2])
def fit_func(x, *p):
ncomp = int(len(p)/3)
y = np.zeros(x.shape[0])
for i in range(ncomp):
y = y + gauss(x, p[3*i:3*i+3])
return y
def plot(ax, x, y_exp, param):
par = param.reshape((-1, 3))
for i, y in enumerate(y_exp):
label = "f{}".format(i+1)
p = ax.plot(x, y, ".", ms=4, label=label + " data")
ax.plot(x, gauss(x, par[i]), color=p[0].get_color())
p = ax.plot(x, y_data, ".", label="data", ms=4)
ax.plot(x, fit_func(x, *param), color=p[0].get_color())
ax.legend()
# 実験データの生成
x = np.linspace(-30, 30, 100)
y_exp = np.empty((0, x.shape[0]))
y_exp = np.concatenate((y_exp, [gauss(x, [1, 3, 50])]), axis=0)
y_exp = np.concatenate((y_exp, [gauss(x, [-0.2, 6, 40])]), axis=0)
# fitting
y_data = np.sum(y_exp, axis=0)
param = curve_fit(fit_func, x, y_data, p0=p0)[0]
param_b = curve_fit(fit_func, x, y_data, p0=p0, bounds=bounds)[0]
fig, ax = plt.subplots(ncols=2, figsize=(14, 6))
plot(ax[0], x, y_exp, param)
plot(ax[1], x, y_exp, param_b)
ax[0].set_title('no bound')
ax[1].set_title('bound')
fig.savefig("fit.png")
plt.show()